基因组光学图谱技术是一种利用单分子DNA的限制性内切酶图谱生成高分辨率、有序的全基因组限制性内切酶酶切图谱的方法.
2010 年,OpGen 公司面向市场推出了基于光学图谱**技术的自动化的基因组拼接和数据分析系统——Argus™工作站,使研究人员能够在一个工作日内获得待测微生物基因组的高质量全基因组限制性内切酶酶切图谱。该系统主要由Mapcard 加工工作站、光学扫描系统和数据处理工作站等组成,通过限制性内切酶对固定于MapCardDNA surface 区域中的单分子DNA 进行原位切割,使切割后的DNA 片段顺序保持不变。DNA 片段经荧光染料染色后置于荧光显微镜下,采集每个限制性内切酶片段的大小和顺序的信息,信息经转换处理后生成单个DNA 分子的限制性内切酶酶切位点图谱,最后根据全部DNA 分子限制性内切酶酶切位点图谱的相互重叠部分拼接得到全基因组限制性内切酶酶切位点图谱。该系统最初被应用于微生物基因组序列组装、比较基因组学以及菌株分类,目前已经开始在人类和动植物全基因组组装中应用。
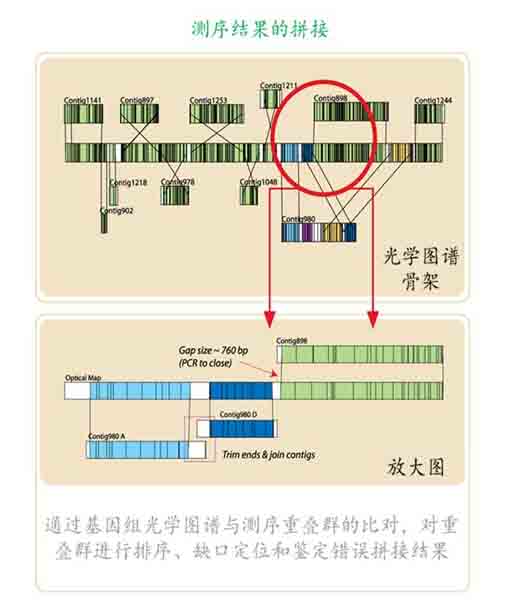
作为对基因组454-Sanger 杂交测序法的一种新的替代技术,光学图谱技术提供了一种全新的微生物基因组测序和拼接策略,虽然该技术不能直接获得基因组DNA 的碱基序列,但它可以提供微生物基因组整体有序的物理框架结构。通过与新一代测序技术的结合,光学图谱技术可以提供基因组绝大多数Contig 的位置和顺序信息,从而辅助基因组的Scaffold 构建,在复杂微生物基因组(多重复序列)的Gap closure 中已经被证明行之有效。
如在嗜线虫致病杆菌(Xenorhabdus nematophila)基因组的Gap closure 过程中,由于含有多达几百个转座子序列以及7 套核糖体RNA 序列(16-23-5S),导致经过4 个月的拼接后还剩余36 个Contig 无法找到Linkage 关系完成拼接,借助于光学图谱技术研究人员通过将嗜线虫致病杆菌高质量的基因组酶切物理图谱(基因组限制性内切酶酶切图谱)与基因组序列模拟酶切图谱(根据测序序列信息计算机模拟所生成基因组In silico 酶切图谱)进行比较,最终将36 个Contig 定位,并且通过比较图谱之间的差异还发现几处由于新一代测序技术自身缺陷(读长短、碱基插入和缺失)所造成的Contig 本身拼接错误,经过纠正,在一个月内完成了嗜线虫致病杆菌基因组的Gap closure。
除了基因组序列模拟的酶切图谱,Argus™系统还可以整合其它一些序列的酶切物理图谱信息( 文库克隆序列、Gap closure PCR 产物以及Paired-End 序列等)用于辅助微生物基因组的Gap closure 工作。
技术流程